之前用于去除嵌合体的工具包括:CHIMERA_CHECK(Maidak, et al., 1999), Pintail (Ashelford, et al., 2005),Mallard(Ashelford, et al., 2006), Bellerophon(Huber, et al., 2004), Chi-meraChecker (Nilsson, et al., 2010),ChimeraSlayer (Haas, et al.,2011) and Perseus (Quince, et al., 2011)。Pintail和Mallard是基于可信赖的数据的比对。CHIMERA_CHECK是ITS标记的方法,用的是BLAST。ChimeraSlayer跟不好嵌合体的数据库进行多重比对。Perseus是454中的用于去除嵌合体的,从头的方法。
Uchime既可以基于不含嵌合体的序列比对,又有de novo模型。
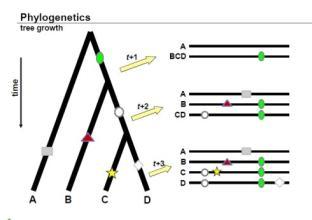
上图是嵌合体的3种比对模型,其中Q是输入序列,而A,B则为父链
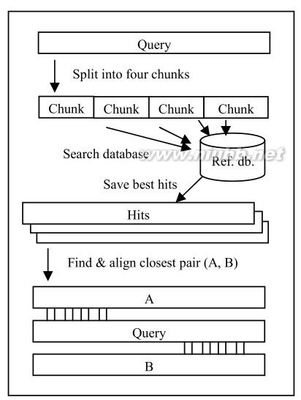
这个图是uchime序列比对的算法。输入序列被切割成4个没有重叠的部分(chunk),每一个用来去比对数据库的序列,每一个chunk匹配上的数据库中的序列被标记出来,通过计算相似性最近的两条父链被发现。A three-way chimeric alignment is constructed, anda chimera is reported if its score (Eq. 2) exceeds a presetthreshold.
从头模型,跟Perseus的原理差不多,因为PCR扩增过程中父链要比嵌合体的循环的次数多,所以丰度上会有变化。
uchime_ref根据序列比对来去除chimera
/sam/qiime/Uparse/usearch -uchime_ref lib1/seqs.fna -db/sam/qiime/Uparse/gold.fasta -uchimeout results.uchime -uchimealns alnfile -chimeras ch.fasta -nonchimeras good.fasta -strandplus
uchime_ref
-db 16S gene ,推荐的数据为gold database (不建议用大的16S database likeGreengenes,下载地址为http://drive5.com/uchime/uchime_download.html),针对ITS,推荐使用数据库UNITE。
‑strand 必须有,而且后面跟plus,意思是数据库必须。This means that the database must be oriented onthe same strand as the query sequences (or contain both forward andreverse-complemented reference sequences).
生成文件:
-Uchimeouthttp://drive5.com/usearch/manual/uchimeout.html
UchimealnsHuman-readable 3-way alignments of query to putativeparents.
ChimerasChimera序列
Nonchimeras非Chimera序列
Qiime也融合了uchime,脚本为identify_chimeric_seqs.py
identify_chimeric_seqs.py -m usearch61 -i lib1/seqs.fna -r/sam/qiime/Uparse/gold.fasta -ousearch61_chimera_checking/ --non_chimeras_retentionintersection
参考资料:
UPARSE pipelinehttp://drive5.com/usearch/manual/uparse_pipeline.html
uchime_ref command http://drive5.com/usearch/manual/uchime_ref.html
usearch manual http://drive5.com/usearch/manual/
文献:UCHIME improvessensitivity and speed of chimera detection
 爱华网
爱华网