根据蛋白质保守区设计简并引物扩增出部分片段是克隆蛋白质家族基因的首要的一步,这儿我发现了一个好工具,与大家共享,可是我还不太会用,如果有用得很熟的不知道是否可以说一说使用的方法和体会。
http://blocks.fhcrc.org/blocks/codehop.html
我用过。
网址是:http://blocks.fhcrc.org/blocks/make_blocks.html,进入后,
1. 框中填入所有(或部分)同源蛋白的氨基酸序列(FASTA格式,这可以从基因银行中找到),然后击“makeblock”,
2.出现“ Making your blocks, please wait...
Your results will be available for approximately 24 hours here.”,击“here”
3.出现"Block Maker Results" sucha as A B C etc.下面是很多Blocks.
4. If you click Map 旁的"Graphical map",进入后,Sequence一项中,一直线上颜色相同的框代表保守的Domain(Its number vary amongdifferent sequence, 2.3.4 or more).
5. 如果你是设计引物,击Primers旁 的 [CODEHOP],进入后,里面有许多选项,- degeneracy[default=128]:
- strictness [default=0.0]:
- temperature [default=60.0]:
- poly-nuc [default=5]:
这些选项你可以增加或减少(根据你的需要),
Then click"Look for primer"
6 进去后,就是软件设计好的兼并引物。 有的Block,可能没有引物( No suggested primersfound.有的Block, 上面是amino acid sequence, 下面是相对应的引物。每个Block都有Forwardand Reverse primer.
If there is no primer, you can increase the strictness,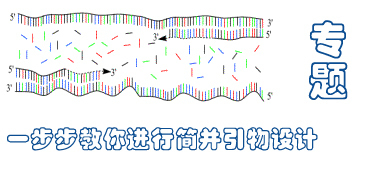
这个程序设计的引物由5‘的clamp(specific)and 3'end的core(degenerated)序列。我用这个程序设计引物成功分离出基因片断。但是, 我不完全用它的序列,稍微改动:
1, 我要查文章, 找到该 同源基因的保守Domain(往往这些domain也是block ),从这些block种找引物。
2。3‘core 序列只有11个nucleotide sequence is degenrated, 我往往查一些基因的序列,再3‘core前面的一个amino acid sequence 也让他兼并。
3。 5‘clamp 序列太长,我都去掉一些, 让整个引物的长度为22-24个碱基就可。
4。由于该引物有clamp and core组成, 有一篇文章, 介绍PCR programme which iscomplex.Now I do not have the article on hand. If you want , pleasetell me(quanzi@hawaii.edu) . But, after I adjust the primer, I usethe norman programe. For example, 94 degree 50s, 50-56degree 50s,72degree30s-2mins. 30-35cycles.
The article is"Consensus-Degenerated Hybrid oligonucleotideprimer for amplification of priming glycosyltransferase genes ofthe exopolysaccharide locus in strains of the lactobacillus caseigroup",magazine is Applied and Environmental Microbiology,publishedin June 2003, volume69,p 3299-3307.
 爱华网
爱华网