江西农业大学学报 2016,38(3):565-572ActaAgriculturaeUniversitatisJiangxiensis
(3):565-572.DOI:10.13836/j.jjau.2016081http://xuebao.jxau.edu.cn胡颂平,吴云花,邹国兴,等.CRISPR/Cas9介导基因组编辑技术在植物基因中的研究进展[J].江西农业大学学报,2016,38
CRISPR/Cas9介导基因组编辑技术
在植物基因中的研究进展
胡颂平1,吴云花1,邹国兴2,聂元元2,杜 鹃1,
毛伟伟1,余 霞1,张 琳1∗
江西南昌330200)(1.江西农业大学生物科学与工程学院,江西南昌330045;2.江西省超级水稻研究发展中心/水稻国家工程实验室,
摘要:CRISPR/Cas9介导的基因组编辑技术是在DNA双链的特定位置形成双链断裂,然后通过同源重组或非同源末端连接方式进行修复,造成基因组碱基局部缺失或插入而引起基因突变,它具有操作简单、突变效率高等优势。笔者归纳了CRISPR/Cas9系统的基本结构、分类及其在植物基因中的研究进展和未来的发展方向。关键词:CRISPR/Cas9;基因组;同源重组;基因突变
中图分类号:Q78 文献标志码:A 文章编号:1000-2286(2016)03-0565-08
TheProgressofGenomicEditingTechnologyMediatedbyCRISPR/Cas9inPlants
HUSong-ping1,WUYun-hua1,ZOUGuo-xing2,NIEYuan-yuan2,DUJuan1,MAOWei-wei1,YUXia1,ZHANGLin1∗
330200,China)JiangxiSuper-riceResearchandDevelopmentCenter/NationalEngineeringLaboratoryforRice,Nanchang
Abstract:CRISPR/Cas9-mediatedgenomiceditingcouldgeneratedoublestrandedbreaksinspecial(1.CollegeofBioscienceandBioengineering,JiangxiAgriculturalUniversity,Nanchang330045,China;2.sites,whichcouldberepairedbyhomologousrecombinationornon-homologousendjoining,anditresultedinbasedeletionorinsertioningenome.TheadvantagesoftheCRISPR/Cas9aresimpleoperationandhigheffi-ciencyofmutation.ThearticledescribesthebasicstructureandclassificationoftheCRISPR/Cas9anditsde-velopmentandprogressinplants.Keywords:CRISPR/Cas9;genome;homologousrecombination;genemutation
近年来,基因组编辑技术为生物学研究开辟了新的研究领域,它是研究基因功能和基因治疗的重要方法。早期,研究人员利用同源重组(Homologousrecombination,HR)的方法对基因进行改造修饰, 收稿日期:2015-12-24 修回日期:2016-01-14
(2010GZN0038、S2016ZRZDB0043)基金项目:国家自然科学基金项目(31260314)、国家留学基金项目(201408360034)和江西省自然科学基金项目
ProjectsupportedbyNationalNaturalScienceFundationofChina(31260314),ChinaScholarshipCouncil
作者简介:胡颂平(1969—),男,教授,主要从事植物分子育种与植物细胞工程研究,sp6974@163.com;∗通信作者:
张琳,zhanglin06@163.com。(201408360034)andNaturalScienceFundationofJiangxi(2010GZN0038,S2016ZRZDB0043)
·566· 江西农业大学学报第38卷发现这种方法的精确度很高,但其打靶效率极低(10-6~10-9)[1]。随着锌指核酸酶(zinc-fingernuclea-ses,ZFNs)[2-3]、类转录因子效应物核酸酶(transcriptionactivator-likeeffectornucleases,TALENs)[4-7]和巨核酶技术(meganucleases)[8]等DNA核酸酶的发现和合成改变了这一现状,这些DNA核酸酶能够在基因组特定的位置形成双链断裂(doublestrandbroken,DSB),然后通过非同源末端连接(non-homologousendjoining,NHEJ)和同源重组进行修复,造成基因组局部碱基的缺失或者插入而起到基因编辑的作用[9]。其中巨核酶是由核酸酶和DNA的结合位点组成,ZFNs和TALENs是由DNA结合位点与FokI酶相互结合组成。虽然ZFNs、TALENs和巨核酶技术能够使基因在特定的位点产生突变,但是具有打靶位点少、周期长、载体结构复杂、所需时间长等缺点[10]。2013年,另一种新的具有代表意义的基因编辑技术CPISPR/Cas9技术诞生,它的工作机理与DNA核酸酶类似,在DNA的特异位点形成双键断裂,再由同源重组或非同源末端连接对DNA进行修复,它具有载体结构简单、效率高、打靶位点多、花费低等特点[10-11]。这种技术已广泛用于人类、小鼠、细菌、酵母菌、斑马鱼、拟南芥、水稻、小麦、果蝇、玉米、烟草等基因组编辑[12-21]。1 CRISPR/Cas系统的基本结构和类型
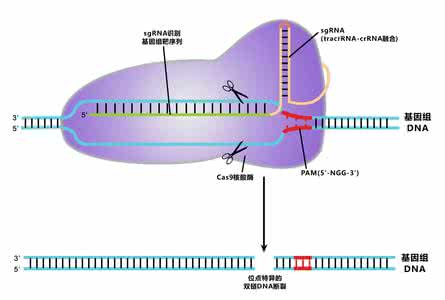
CRISPR/Cas系统主要由成簇规律的短回文序列(clusteredregularlyinterspacedshortpalindromicre-peats,CRISPR)和编码核酸酶的Cas基因组成,成簇规律的短回文序列主要由短小的正向重复序列和完全不同的间隔序列交替组成。CRISPR/Cas系统主要存在于细菌和古生菌中,CRISPR/Cas系统中的重72bp[22-24]。科学家研究发现,在已研究调查的细菌基因组中有40%的基因组中存在CRISPR/Cas位点[25]。不同物种CRISPR位点的数量不同,在已检测到CRISPR/Cas系统的细菌中,有1/2的细菌含有多个CRISPR位点,并且平均每个位点有27个重复序列,重复序列的平均长度为32bp[26]。在正向重复序列的上游存在长度约为200bp的前导序列,它主要用于启动CRISPR序列的转录[27]。在CRISPR或者病毒进行降解。附近存在多种类型的Cas基因。多种Cas基因编码的Cas蛋白能够形成复合体,对外源入侵的噬菌体1987年,研究人员在大肠杆菌中发现长度为29nt(nucleotide)的重复序列被32nt的非重复序列分复序列是一段长为21~47bp的回文重复序列,间隔序列的长度与重复序列的长度相似为21~隔开的CRISPR序列[28]。2002年,科学家对CRISPR序列正式进行命名,同时在CRISPR的附近发现一些与重复序列有关能够编码Cas蛋白的编码基因[27]。随后,研究人员发现CRISPR/Cas系统有3种类型,分别为TypeI、TypeII、TypeIII。而且3种类型的CRISPR/Cas系统主要由Cas基因家族和CRISPR序列元件组成[29]。TypeI系统在细菌和古生菌中都有发现,它包含6个亚型,所有的亚型中都有Cas7、Cas5、Cas3蛋白[30]。Cas3蛋白是TypeI系统中的标志性蛋白,它有一个磷酸水解酶结合位点和一个解旋酶结合位点,并且它能够和不同Cas蛋白形成复合体与crRNA(CRISPR-derivedRNA)进行结合形成CASCADE(CRISPRassociatedcomplexforantivirusdefense)复合体,然后,通过原间隔序列(protospacer)下游保守序列(protospaceradjacentmotifs,PAM)来识别DNA靶序列,使DNA的结构发生变化[31-34]。TypeI系统中PAM位点一般为2~3个碱基,并且在不同的亚型中序列的长度不同。TypeII系统只在细菌中有发现,这个系统有一个多功能的Cas9蛋白。Cas9蛋白有2个特征性的核酸酶结构域分别是RuvC和HNH,其中HNH是一个单一的核酸酶结构域,RuvC包括3个子结构域RuvCI、RuvCII、RuvCIII。RuvCItracrRNA-Pre-crRNA在RNaseIII的作用下完成pre-RNA的成熟形成crRNA[35-36]。TypeII系统中proto-在Cas9蛋白的N端,而RuvCIII和RuvCII与HNH侧面相接在蛋白的中央。Cas9蛋白能够促进tracrRNA(trans-activatingcrRNA)与CRISPR重复序列前导区转录形成的pre-crRNA进行配对,使spacer序列下游的PAM是一个为5个碱基的保守序列(NGGNG),tracRNA与crRNA配对形成的二聚体通过对PAM保守序列的识别,引导Cas9蛋白在距离PAM位点3~8个碱基的位置对目标序列进行剪切。TypeIII系统主要存在于古生菌中,它包括TypeIIIA、TypeIIIB2个亚型[31-32]。Cas6蛋白和Cas10蛋白是TypeIII系统的特征性蛋白。TypeIIIA系统包括5种Cas蛋白,在此系统中不需要特异性的PAM保守序列,并且与crRNA序列5′端8个核苷酸互补的序列不能作为靶序列[37-38]。在TypeIIIB中Cas6蛋
第3期胡颂平等:CRISPR/Cas9介导基因组编辑技术在植物基因中的研究进展·567·白是对抵抗外源DNA必不可少的一部分。并且crRNA序列5′端的8个核苷酸是6种Cas蛋白形成复合体的嵌合点。TypeIII系统同样是通过crRNA与多种Cas蛋白形成复合体对外源DNA进行剪切。切酶主要来自酿脓链球菌的CRISPR/Cas系统[39]。2012年,研究人员发现,crRNA在试管中也能引导Cas9蛋白对目标序列进行剪切,并且在同一年,一条由crRNA和tracrRNA嵌合而成的单链引导RNA(sgRNA)诞生[36,39]。sgRNA合成使CRISPR/Cas9系统不需要RaseIII的作用,直接就能引导Cas9对目标序列的剪切使DNA双键断裂,并以同源重组或非同源末端连接等方式对DNA进行修复。异源体sgRNA引导Cas9对目标序列的剪切比crRNA更加高效。2013年,CRISPR/Cas9系统成功地用于哺乳动物基因编辑[40]。目前这项基因编辑技术已广泛地运用于多种动植物基因中。现在广泛用于基因编辑技术的CRISPR/Cas9系统是根据TypeII系统进行改造而来的,Cas9核酸内2 CRISPR/Cas系统的免疫机制
细菌和古生菌中CRISPR/Cas系统主要是为了抵御外界噬菌体或病毒的侵入而形成的自身免疫系统。CRISPR/Cas系统的免疫机制主要为以下几个方面。首先,当病毒或噬菌体侵入细菌时,细菌中CRIPSR/Cas系统中的外源DNA特定的原间隔序列(protospacer)和PAM序列对外源DNA进行识别,并毒再一次侵染宿主菌时,CRISPR/Cas系统的前导区会转录形成pre-crRNA(precursor-crRNA)。此转录必须同时存在启动子元件和Cas蛋白结合位点的情况下才能发生[43-44]。Pre-crRNA在核酸内切酶和tracrRNA的加工下形成成熟的crRNA,成熟的crRNA包括间隔序列、5′端羟基的重复序列和2~3个磷酸化循环末端[45-46]。crRNA与Cas蛋白结合形成复合体对外源入侵的噬菌体或者病毒进行攻击而致使核酸降解[47],而达到对自身免疫的作用。且宿主菌将外源DNA重新整合进入CRISPR/Cas系统中的间隔序列[41-42],形成“记忆”。当噬菌体或病3 CRISPR/Cas9在植物中的研究进展
2013年,Feng等[13]在sgRNA载体上分别连接AtU6-26启动和OsU6-2启动子来调控sgRNA在不同植物中的瞬时表达。发现带有AtU6-26启动子的sgRNA适合用于拟南芥等双子叶植物基因编辑,带有OsU6-2启动子的sgRNA适合用于水稻等单子叶植物基因编辑。目前,CRISPR/Cas9基因编辑技术已经3.1 CRISPR/Cas9系统在单子叶植物中的研究进展广泛用于各种植物的基因编辑。
2013年,Xie等[48]构建带有CaMV35S启动子CAS9载体和带有U3或U6启动子的sgRNA表达载体,并利用这套CRISPR/Cas9系统靶向水稻的OsMPK5基因。研究发现,利用原生质体瞬时表达转化载体,在转化后18h,其转化率达到了60%,在36~72h其转化达到90%。进行非同源末端连接修复发生缺失突变和插入突变的比例为1∶1,其中缺失突变的片段大小为3~14bp,插入突变的片段大小为42~195bp。对转基因植株进行检测发现基因突变的效率为3%~8%。2014年,Zhang等[49]利用CRISPR/Cas9编辑技术对水稻的11个不同基因进行基因编辑,发现在T0代植株中突变植株的平均概率为44%,这些突变植株中有4%的植株是双等位突变,并且这些突变植株都能稳定遗传给下一代。对突变植株的DNA序列进行测序发现这些植株中极少数存在脱靶的现象。2013年,研究人员[50]设计了sgRNA和双链crRNA-tracrRNA(dual-crRNA:tracrRNAs)对水稻内源基因进行研究,探索这2种RNA对2个外显子和控制分蘖夹角的LAZY1基因的第3个外显子设计目标序列,利用CRISPR/Cas基因编辑技术分别对这2个外显子进行编辑,利用农杆菌转化水稻的愈伤组织。CAO1基因在苗期开始表现功能缺失,叶片呈现淡绿色。在水稻的分蘖时期,LAZY1基因表现功能缺失,分蘖性状为散射状。对sgRNA引导的T0代突变植株进行统计发现2个基因的突变率分别达到了13.3%和50%,其中T1代植株中为突变植株的概率分别是83.3%和91.6%,而dual-crRNA:tracrRNAs引导的2个基因突变率只有4.44%,由此说明,在产生内源基因的定点突变基因方面sgRNA引导的系统比dual-crRNA:tracrRNAs引导的系统效率更高。2015年,Mikami等[51]将Cas9和sgRNA两个表达组件转入一个双运表达载体中,对控制水稻叶色的PDS(phytoenedesaturase)基因和控制叶片垂直度的DL(droppingleaf)基因进行编辑,检测在一水稻的内源基因编辑效率的高低。在控制水稻叶绿素含量的CAO1(chlorophyllAoxygenase1)基因的第
·568· 江西农业大学学报第38卷个表达载体中CRISPR/Cas系统的打靶效率。研究发现,PDS基因转化愈伤组织的转化率为90.4%。愈伤组织再生成为突变植株的概率为42.1%,其中发生等位双突变的植株叶片呈现白化的现象。DL基因中的4个靶点发生突变的最高频率为93%,愈伤组织的转化率为100%,发生等位双突变的植株出现叶片严重下垂的现象。由此说明Cas9与sgRNA在同一个表达载体中的CRISPR/Cas9系统能够高效地对基因进行编辑。2014年,Xu等[52]利用CRISPR/Cas9编辑技术针对水稻抗除草剂BEL(bentazonsen-sitivelethal)基因的第2个外显子设计靶序列,并且利用农杆菌介导转化植物愈伤组织。对转基因植株进行测序分析发现,在90株转基因植株中有15株发生碱基的缺失或碱基替代,并且筛选出一株双等位(在第17个碱基和第14个碱基处都发生碱基缺失)突变株系。对与双突变型植株处于同样生长期野生型植株同时喷射等量的除草剂,发现野生型植株的叶子没有变化,而双突变植株的叶子为黄色并且有严重褶皱。Max等[53]利用CRISPR/Cas9系统对水稻基因设计46个靶位点,检测到这些靶点的平均突变频率为85.4%,主要的突变类型是双等位突变和同源突变。Xing等[54]以玉米ZmHKT1基因作为靶基因,在靶基因上设置2个不同的靶点,检测CRISPR/Cas9系统在单子叶植物中的突变效率。研究结果发现,每个靶点平均发生突变的转基因植株占所得转基因植株的60%。Upadhyay等[18]针对小麦inox(inositoloxygenase)基因和PDS(phytoenedesaturase)基因设22%,其中inox基因发生缺失突变最大的片段是24bp,插入突变最大的片段是22bp,并且在距离研究人员用一个原间隔序列的PAM同时对inox基因和PDS基因进行打靶,研究结果发现,PDS基因发3.2 CRISPR/Cas9在双子叶植物中的研究进展生突变的效率为10%,inox基因发生突变的效率为8%[18]。计靶序列,利用CRISPR/Cas9技术对基因进行编辑,在原生质体中检测到两个基因突变效率为18%~PAM2~3个碱基处发生插入或缺失突变。为了检测CRISPE/Cas9系统同时打靶两个不同基因的效率,
2013年,Feng等[13]利用CRISPR/Cas9系统对拟南芥耐油菜素类固醇BRIL基因和赤霉素GAI基因进行基因编辑,共获得50株T1代转基因植株和20株T0代转基因植株,其中,T1代转基因植株在拟南芥早期生长阶段就表现出多种生长表型,以BRIL基因为靶序列的转基因植株在T1代有50%表现出生长阻滞、叶片卷曲,GAI转基因植株在T1代中有四分之一的植株外形矮小。第2年,Feng等[55]又对拟南芥的7个基因共12个靶点进行编辑,在T1代、T2代、T3代转基因植株中检测的突变效率分别为传到T3代植株中。Xing等[54]对p2gR-TRI-A和p2gR-TRI-B2个载体构建不同的sgRNA(T1A-TC和T1B-TC),这2个载体同时打靶控制拟南芥表面毛状物的两个基因(TRY和CPC),研究发现,以T1A-TCT1代植株中只有少于10%的植株是所需要的转基因植株。Jiang等[56]构建含有U6启动子CRISPR/TRI-A载体的T1代转基因植株有70%的株系的叶片表面都是高密度的毛状物,而p2gR-TRI-B载体的Cas9的双运载体对无功能的GFP基因进行打靶使无功能GFP基因突变为有功能的GFP,对T0代转基因植株进行潮霉素筛选和PCR检测,从稳定遗传到T1代的拟南芥突变植株叶片中可以观察到绿色荧光信号,而且其突变性状能够稳定的遗传到T2代和T3代植株中。Hyum等[57]针对拟南芥的FT(Flow-eringlocusT)基因和SPL-4(Squamosapromoterbindingprotein-like4)基因进行靶点设计,在T1代转基因植株中均检测到突变位点。FT突变植株在T1代表现出开花延迟,这些突变植株中90%的植株表现单碱基插入或小片段缺失的突变类型。Wang等[58]从8个卵细胞特有的启动子与2个不同的终止子(pi-sumsativumrbcSE9、agrobacteriumnos)形成12个组合中筛选最适合用于CRISPR/Cas9系统编辑的终止子和启动子。利用农杆菌转化拟南芥胚胎形成的愈伤组织。研究发现,采用2个启动子融合的CRISPR/Cas9系统的打靶效率高于单个启动子的效率,其纯合突变或双等位突变效率达到38.8%。Yin等[59]构建以烟草PDS为靶基因、卷叶虫病毒和TDNA载体为基础含有U6启动子和gRNA的71.2%、58.3%、79.4%。其中T2代突变植株中有22%的纯合突变植株,并且这些纯合突变都能稳定的遗为sgRNA的载体与目标基因之间发生1个错配,T1B-TC与目标基因之间发生2个错配。靶向p2gR-病毒载体(pCVA-gRNA::NbPDS/pCVB),通过对Cas9表达载体能够高效表达的转基因烟草(KQ334)叶片注射病毒载体使病毒侵染到植物细胞,并且通过自身的复制使gRNA表达。最后Cas9蛋白在gRNA的引导下对靶基因进行剪切而达到基因编辑的目的(VIGE)。研究发现,有30%KQ334植株叶片在连续注射pCVA-gRNA::NbPDS/pCVB的第3个星期出现了光学漂白的现象,在12个星期时可以观察到
第3期胡颂平等:CRISPR/Cas9介导基因组编辑技术在植物基因中的研究进展·569·明显的现象,而只注射了pCVA-scaffold/pCVB载体的叶片并没有出现该现象。对具有明显光学漂白的叶片进行PCR和测序检测发现PDS基因已经发生突变,有1~8bp的碱基发生缺失,1bp的碱基发生插入。研究人员以控制烟草叶色的NbIspH基因为靶序列设计pCVA-gRNA::NbIspH/pCVB载体进一步验证VIGE编辑技术,有50%KQ334的植株叶片在连续注射pCVA-gRNA::NbIspH/pCVB载体后出现光学漂白现象。运用PCR检测光学漂白现象明显的植株,发现基因突变效率为75%。Gao等[60]构建其突变植株的叶片呈现黄色,NtPDR6基因的突变频率87.5%,其突变植株的枝条比野生型多。CRISPR/Cas9系统诱导烟草NtPDS基因和NtPDR6基因突变。其中,NtPDS基因的突变频率为81.8%,4 CRISPR/Cas9系统面临的挑战和未来的发展
尽管CRISPR/Cas9已经广泛地运用于各种生物的基因编辑,但是,系统中某些结构的作用机制还4.1 提高CRISPR/Cas9系统的特异结合能力和效率不明确,而且某些应用还需要进一步的提高。
CRISPR/Cas9系统的一个关键需要是最大限度地减小脱靶的影响。目前,也有几种方法用于减少脱靶的影响,如:缩短gRNA的长度,使用双切口酶,运用dcas9-FokI复合体等,然而一个gRNA序列可能有很多的脱靶位点,每个位点都需要实验数据分析其脱靶的影响。近年来也有许多检测脱靶方法生成。如Kim等[61]运用Digenome-seq方法分析基因脱靶的影响,Frock等[62]运用HTGTS(Genome-wide的基因编辑,研究者也希望有更多的方法能够用于检测基因脱靶和减少脱靶效率。translocationsequencing)方法对DNA双链的断裂进行检测。随着CRISPR/Cas9系统用于越来越多生物
众所周知,高效的基因编辑的特异性位点核酸酶的选用是依据基因组不同遗传组合和不同特异性位点的表观遗传状态来选择的。例如,每条DNA的可编辑性都有一一对应的转录因子结合和最新的Cas9蛋白结合[63-64],各种各样的组蛋白修饰也都有一一对应的转录因子和染色体结构[65-67]。由于复杂催化活性和高效的CRISPR/Cas9系统的影响都没有深入的理解。而对CRISPR/Cas9系统机制深入了4.2 扩大CRISPR/Cas9系统的适用范围。
目前,基因编辑技术主要用于内源基因的功能性缺失突变和修饰,而以CRISPR为基础的基因编辑技术主要是专注于Cas9核酸酶的发展。在CRISPR/Cas9系统类型和功能的多样性等方面的研究还是比较欠缺,只有少量的研究报道过这方面相关的某些研究。2012年,Hale等[43]从强烈火球菌和硫磺矿硫化叶菌中鉴定出的CasRAMP蛋白能够与sgRNA形成复合物对目标RNA进行剪切。CasRAMP蛋白的剪切位点在目标序列的5′端,目标序列主要由6个碱基的中心区域和另外13个碱基组成。2013年,Chen等[68]通过对trascrRNA的茎环结构增加一个A-U环,成功地将CRISPR/Cas9系统高效地运用于基因抑制和对基因位点的分类。此外,Konermann等[69]和Zalatan等[70]发现sgRNA中不同的茎环结构通过结合不同的转录因子来起到激活基因和抑制基因的作用。并且,Zalatan等[70]发现带有不同茎环的sgRNA靶向不同的基因位点,同一个dCas蛋白在不同的位点执行的功能不同。随着科学技术的不断发展基因编辑技术的应用越来越广泛,CRISPR/Cas9介导的基因编辑的适用范围也需要不断的拓展。解能够提高基因编辑方面的准确性和效率。的遗传序列、表观遗传修饰和不同的遗传位点交叉在不同的细胞中,至今研究人员对位点的结合机制,5 展望
传统的基因编辑技术如基因枪法、花粉管道法和农杆菌介导T-DNA插入法,将外源基因随机地插入到植物基因组中,进而获得相应性状的植株。由于随机向基因组插入目标基因,对宿主基因造成的伤害不能够预计,它可能导致内源基因受损或外源基因沉默。由于植株基因组中自发地发生同源重组的概率很低,想要获得特定位点发生基因突变的目标性状概率更低。但是,当植物基因组中的双链发生断裂时,基因发生同源重组和非末端同源重组的概率大大地提高。而现在广泛用于基因编辑的CRISPR/Cas9系统就是利用DNA在特定位点形成双链的断裂,再在同源重组或非末端同源重组的作用下,造成碱基的插入或者缺失使基因组发生突变。相较于传统的基因编辑技术,CRISPR/Cas9介导的基因组编辑技术具有操作简单,效率高,适用范围广、花费低等优点,它是基因工程研究方面的一个新思路和新方
·570· 江西农业大学学报第38卷法,它将有可能在生物技术领域上产生积极的作用。
断改进和提高,例如,如何解除PAM序列的限制、如何最大范围地减少脱靶突变、如何构建在不同物种中通用的Cas9与sgRNA体系,怎样提高同源重组修复效率等。随着基因工程不断深入地研究,CRISPR/Cas9系统在动植物基因改造、作物遗传育种、基因转录调控、表观遗传调控、动植物品种改良等方面将有更广阔的发展前景。
参考文献:
[1]CapecchiMR.Alteringthegenomebyhomologousrecombination[J].Science,1989,244(4910):1288-1292.
NatBiotechnol,2007,25(7):778-785.[2]MillerJC,HolmesMC,WangJ,etal.Animprovedzinc-fingernucleasearchitectureforhighlyspecificgenomeediting[J].[3]UrnovFD,MillerJC,LeeYL,etal.Highlyefficientendogenoushumangenecorrectionusingdesignedzinc-fingernuclea-[4]ChristianM,CermakT,DoyleEL,etal.TargetingDNAdouble-strandbreakswithTALeffectornucleases[J].Genetics,[5]MillerJC,TanS,QiaoG,etal.ATALEnucleasearchitectureforefficientgenomeediting[J].NatBiotechnol,2011,29[6]BochJ,ScholzeH,SchornackS,etal.BreakingthecodeofDNAbindingspecificityofTAL-typeIIIeffectors[J].Science,[7]MoscouMJ,BogdanoveAJ.AsimpleciphergovernsDNArecognitionbyTALeffectors[J].Science,2009,326(5959):[8]SmithJ,GrizotS,ArnouldS,etal,Acombinatorialapproachtocreateartificialhomingendonucleasescleavingchosense-[9]HsuPD,LanderES,ZhangF.DevelopmentandapplicationsofCRISPR-Cas9forgenomeengineering[J].Cell,2014,157[10]ShanQ,WangY,LiJ,etal.GenomeeditinginriceandwheatusingtheCRISPR/Cassystem[J].NatProtoc,2014,9(10):[11]MussolinoC,CathomenT.RNAguidesgenomeengineering[J].NatBiotechnol,2013,31(3):208-209.
tobacco,sorghumandrice[J].NucleicAcidsRes,2013,41(20):188.
(10):1229-1232.2395-2410.[12]JiangW,ZhouH,BiH,etal.DemonstrationofCRISPR/Cas9/sgRNA-mediatedtargetedgenemodificationinArabidopsis,[13]FengZ,ZhangB,DingW,etal.EfficientgenomeeditinginplantsusingaCRISPR/Cassystem[J].CellRes,2013,23[14]HwangWY,FuY,ReyonD,etal.EfficientgenomeeditinginzebrafishusingaCRISPR-Cassystem[J].NatBiotechnol,[15]ChangN,SunC,GaoL,etal.GenomeeditingwithRNA-guidedCas9nucleaseinzebrafishembryos[J].CellRes,2013,23[16]BassettAR,TibbitC,PontingCP,etal.HighlyefficienttargetedmutagenesisofdrosophilawiththeCRISPR/Cas9system[17]LiD,QiuZ,ShaoY,etal.HeritablegenetargetinginthemouseandratusingaCRISPR-Cassystem[J].NatBiotechnol,[18]UpadhyaySK,KumarJ,AlokA,etal.RNA-guidedgenomeeditingfortargetgenemutationsinwheat[J].G3(Bethesda),[19]ChoSW,KimS,KimJM,etal.TargetedgenomeengineeringinhumancellswiththeCas9RNA-guidedendonuclease[J].[20]ShanQ,WangY,LiJ,etal.TargetedgenomemodificationofcropplantsusingaCRISPR-Cassystem[J].NatBiotechnol,[21]LiangZ,ZhangK,ChenK,etal.TargetedmutagenesisinZeamaysusingTALENsandtheCRISPR/Cassystem[J].JGenet[22]NakataA,AmemuraM,MakinoK.UnusualnucleotidearrangementwithrepeatedsequencesintheEscherichiacoliK-12Genomics,2014,41(2):63-68.2013,31(8):686-688.NatBiotechnol,2013,31(3):230-232.2013,3(12):2233-2238.2013,31(8):681-683.[J].CellRep,2013,4(1):220-228.(4):465-472.2013,31(3):227-229.(6):1262-1278.quences[J].NucleicAcidsRes,2006,34(22):149.1501.2009,326(5959):1509-1512.(2):143-148.2010,186(2):757-761.ses[J].Nature,2005,435(7042):646-651.CRISPR/Cas9介导的基因编辑技术在基因编辑应用方面属于一个初始阶段,在很多方面还需要不
第3期胡颂平等:CRISPR/Cas9介导基因组编辑技术在植物基因中的研究进展·571·[23]MojicaFJ,FerrerC,JuezG,etal.Longstretchesofshorttandemrepeatsarepresentinthelargestrepliconsofthearchaea
(1):85-93.chromosome[J].JBacteriol,1989,171(6):3553-3556.HaloferaxmediterraneiandHaloferaxvolcaniiandcouldbeinvolvedinrepliconpartitioning[J].MolMicrobiol,1995,17[24]MojicaFJ,Diez-VillasenorC,SoriaE,etal.Biologicalsignificanceofafamilyofregularlyspacedrepeatsinthegenomesof[25]GoddeJS,BickertonA.TherepetitiveDNAelementscalledCRISPRsandtheirassociatedgenes:evidenceofhorizontal[26]BlandC,RamseyTL,SabreeF,etal.CRISPRrecognitiontool(CRT):atoolforautomaticdetectionofclusteredregularly[27]JansenR,EmbdenJD,GaastraW,etal.IdentificationofgenesthatareassociatedwithDNArepeatsinprokaryotes[J].Molinterspacedpalindromicrepeats[J].BMCBioinformatics,2007,8:209.transferamongprokaryotes[J].JMolEvol,2006,62(6):718-729.archaea,bacteriaandmitochondria[J].MolMicrobiol,2000,36(1):244-246.
[28]IshinoMicrobiol,2002,43(6):1565-1575.
conversionY,ShinagawainEscherichiaH,Makinocoli,andK,etidentificational.Nucleotideofthesequencegeneproduct[J].Journaloftheiapgene,responsibleofBacteriology,1987,169:5429-5433.foralkalinephosphatase
[29]HaftCassubtypesDH,SelengutexistinJ,MongodinprokaryoticEgenomes[J].PLoSF,etal.AguildofComput45CRISPR-associatedBiol,2005,1(6):460.(Cas)proteinfamiliesandmultiple[30]2013,14(7):14518-14531.RichterH,RandauL,PlagensA.ExploitingCRISPR/Cas:interferencemechanismsandapplications[J].
[31]MakarovationofCRISPR-CasKS,Aravindsystems[J].BiolL,WolfYI,etDirect,2011,6:38.al.UnificationofCasproteinfamiliesandasimplescenarioforthe
[32]Makarovaol,2011,9(6):467-477.KS,HaftDH,BarrangouR,etal.EvolutionandclassificationoftheCRISPR-Cassystems[J].Nat
[33]WestraBiol,2012,9(9):1134-1138.ER,NilgesB,vanErpPB,etal.Cascade-mediatedbindingandbendingofnegativelysupercoiled
[34]SinkunasthermophilusT,Gasiunas[J].EMBOG,WaghmareJ,2013,32(3):385-394.SP,etal.InvitroreconstitutionofCascade-mediatedCRISPRimmunity[35]DeltchevaIII[J].Nature,2011,471(7340):602-607.E,ChylinskiK,SharmaCM,etal.CRISPRRNAmaturationbytrans-encodedsmallRNAand
[36]Jinek[J].Science,2012,337(6096):816-821.M,ChylinskiK,FonfaraI,etal.Aprogrammabledual-RNA-guidedDNAendonucleaseinadaptivebacterial[37]MarraffiniGenet,2010,11(3):181-190.LA,SontheimerEJ.CRISPRinterference:RNA-directedadaptiveimmunityinbacteriaandarchaea[J].Nat[38]MarraffiniScience,2008,322(5909):1843-1845.LA,SontheimerEJ.CRISPRinterferencelimitshorizontalgenetransferinstaphylococcibytargeting[39]GasiunasG,BarrangouR,HorvathP,etal.Cas9-crRNAribonucleoproteincomplexmediatesspecificDNA
[40][41]Jinekdaptive
MojicaM,Eastimmunity
FJ,Diez-VillasenorA,Chenginbacteria[J].ProcA,etC,al.RNA-programmedNatlAcadSci,2012,109(39):2579-2586.
Garcia-MartinezJ,genomeetal.Shorteditingmotifinhumansequencescells[J].Elife,2013,2:471.
CRISPRdefencesystem[J].Microbiology,2009,155(PtCRISPR3):733-740.determinethetargetsof
[42]One,2012,7(4):35888.SwartsDC,MosterdC,vanPasselMW,etal.interferencedirectsstrandspecificspaceracquisition[[43]HaleRAMPCmoduleR,MajumdarcomplexS,ElmoretocleaveJ,etRNAs[J].Molal.EssentialfeaturesCell,2012,45(3):292-302.andrationaldesignofCRISPRRNAsthatfunction
[44]LillestolanddynamicRK,Shahproperties[J].MolSA,BruggerMicrobiol,2009,72(1):259-272.K,etal.CRISPRfamiliesofthecrenarchaealgenusSulfolobus:bidirectional
[45]SashitaldoribonucleaseDG,JinekCse3[J].NatM,DoudnaStructJA.AnMolRNA-inducedBiol,2011,18(6):680-687.conformationalchangerequiredforCRISPRRNAcleavage[46]JoreStructMMolM,LundgrenBiol,2011,18(5):529-536.M,vanDuijnE,etal.StructuralbasisforCRISPRRNA-guidedDNArecognitionbyisozymeCRISPR/JMolSci,andevolu-RevMicrobi-DNA[J].RNAStreptococcusfactorRNaseimmunityRevDNA[J].cleavagefora-prokaryoticJ].PLoSwiththeCastranscriptionbytheen-J].NatIntorigininhosttheCascade[
·572·
江西农业大学学报第38卷
[47]WiedenheftB,vanDuijnE,BultemaJB,etal.RNA-guidedcomplexfromabacterialimmunesystemenhancestargetrecog-[48]XieK,YangY.RNA-guidedgenomeeditinginplantsusingaCRISPR-Cassystem[J].MolPlant,2013,6(6):1975-1983.
onegeneration[J].PlantBiotechnolJ,2014,12(6):797-807.1236.
nitionthroughseedsequenceinteractions[J].ProcNatlAcadSci,2011,108(25):10092-10097.
[49]ZhangH,ZhangJ,WeiP,etal.TheCRISPR/Cas9systemproducesspecificandhomozygoustargetedgeneeditinginricein[50]MiaoJ,GuoD,ZhangJ,etal.TargetedmutagenesisinriceusingCRISPR-Cassystem[J].CellRes,2013,23(10):1233-[51]MikamiM,TokiS,EndoM.ComparisonofCRISPR/Cas9expressionconstructsforefficienttargetedmutagenesisinrice[52]XuR,LiH,QinR,etal.Genetargetingusingtheagrobacteriumtumefaciens-mediatedCRISPR-Cassysteminrice[J].Rice[J].PlantMolBiol,2015,88(6):561-572.
[53]Ma(NX,ZhangY),2014,7(1):5.
monocotanddicotQ,Zhuplants[J].MolQ,etal.ArobustPlant,2015,8(8):1274-1284.CRISPR/Cas9[54]Xing2014,14:327.
HL,DongL,WangZP,etal.ACRISPR/Cas9toolkit[55]FengducedZ,MaogenemodificationsY,XuN,etinal.MultigenerationArabidopsis[J].ProcanalysisNatlreveals[56]JiangW,YangB,WeeksDP.EfficientCRISPR/Cas9-mediatedAcad[57]HyunifiedgenesY,KiminJ,ChotheT2SandW,etT3al.Site-directedgenerations[J].PLoSmutagenesisOne,2014,9(6):99225.
[58]WangCRISPRZP,Xing/CassystemHL,DongtogenerateL,etheritableal.Eggcell-specificnullalleles[J].Planta,2015,241(1):271-284.mutantsformultipletargetgenesinArabidopsisinasinglepromoter-controlled[59]Yinting[J].SciK,HanT,LiuRep,2015,5:14926.
G,etal.Ageminivirus-basedguideRNA[60]Gao87(1-2):99-110.
J,WangG,MaS,etal.CRISPR/Cas9-mediatedtargeted[61]KimMethods,2015,12(3):237-243.D,BaeS,ParkJ,etal.Digenome-seq:genome-wide[62]Frockses[J].NatRL,HuBiotechnol,2015,33(2):179-186.J,MeyersRM,etal.Genome-widedetection[63]Kuscuclease[J].NatC,ArslanBiotechnol,2014,32(7):677-683.S,SinghR,etal.Genome-wideanalysis[64]Wutechnol,2014,32(7):670-676.
X,ScottDA,KrizAJ,etal.Genome-widebindingof[65]KundajeenvironmentA,Kyriazopoulou-Panagiotopoulouatregulatoryelements[J].GenomeS,Libbrecht[66]WangRes,2012,22(9):1735-1747.transcriptionJ,Zhuangfactors[J].GenomeJ,IyerS,etal.SequenceRes,2012,22(9):1798-1812.
featuresandchromatin[67]Dunham2012,489(7414):57-74.
I,KundajeA,AldredSF,etal.Anintegrated[68]ChenB,GilbertLA,CiminiBA,etal.Dynamicimaging[69]Konermannsystem[J].Cell,2013,155(7):1479-1491.complex[J].Nature,2015,517(7536):583-588.S,BrighamMD,TrevinoAE,etal.Genome-scale[70]Zalatan[J].Cell,2015,160(1JG,LeeME,Almeida/2):339-350.
R,etal.Engineeringcomplexforconvenient,high-efficiencymultiplexgenomeeditinginformultiplexgenomeeditinginplants[J].BMCPlantBiol,Sci,2014,111(12):4632-4637.theinheritance,specificity,andpatternsofCRISPR/Cas-in-geneeditinginarabidopsisthalianaandinheritanceofmod-Arabidopsisthalianausingdividingtissue-targetedRGENofthegeneration[J].GenomeCRISPRBiol,2015,16:144./Cas9efficientlygenerateshomozygousdeliverysystemforCRISPR/Cas9mediatedplantgenomeedi-mutagenesisinNicotianatabacum[J].PlantMolBiol,2015,ofCRISPR-Cas9off-targeteffectsinhumancells[J].NatofDNAdouble-strandedbreaksinducedbyengineerednuclea-characteristicsofoff-targetsitesboundbytheCas9endonu-CRISPRendonucleaseCas9inmammaliancells[J].NatBio-al.Ubiquitousheterogeneityandasymmetryofthechromatinstructurearoundthegenomicregionsboundby119humanofDNAelementsinthehumangenome[J].Nature,genomiclociinlivinghumancellsbyanoptimizedCRISPR/CastranscriptionalactivationbyanengineeredCRISPR-Cas9synthetictranscriptionalprogramswithCRISPRRNAscaffolds
systeminprofilingrevealstheM,etencyclopediaof
百度搜索“爱华网”,专业资料,生活学习,尽在爱华网
 爱华网
爱华网